Das Konzept von SCREAM-DNP (Specific Cross Relaxation Enhancement by Active Motions)
Häufig stößt NMR auf Grund der geringen Empfindlichkeit an Grenzen. Dies bedeutet, dass die Signalstärke nicht ausreichend ist, um die atomare Struktur zu ermitteln. Durch die moderne Methode der dynamischen Kernspinpolarisation (DNP) kann die Empfindlichkeit von NMR drastisch gesteigert werden. Doch auch mit der Verstärkung durch DNP ist die gezielte Strukturanalyse von bestimmten Biomolekülen sehr schwierig, insbesondere, wenn diese z.B. in ihrem natürlichen Umfeld, d.h. neben vielen anderen zellulären Bestandteilen vorliegen und davon unterschieden werden müssen.
Um dieses Problem zu lösen, setzen wir in unserer Forschung eine Methode ein, die wir SCREAM-DNP (Specific Cross Relaxation Enhancement by Active Motions) genannt haben. Durch den Einsatz methyltragender „Spionmoleküle“ können große Biomoleküle ganz gezielt polarisiert werden, z.B., wenn diese in Form von Liganden spezifisch durch ein Protein- oder RNA-Molekül gebunden werden. Diese Methode ist nicht nur beim Nachweis einer Bindung zwischen einem Liganden und einem Biomolekül äußerst nützlich, sondern ermöglicht auch das Ausfiltern aller spektralen Signale von anderen (nicht mit dem Spionmolekül gebundenen) Bestandteilen.
Das Funktionsprinzip
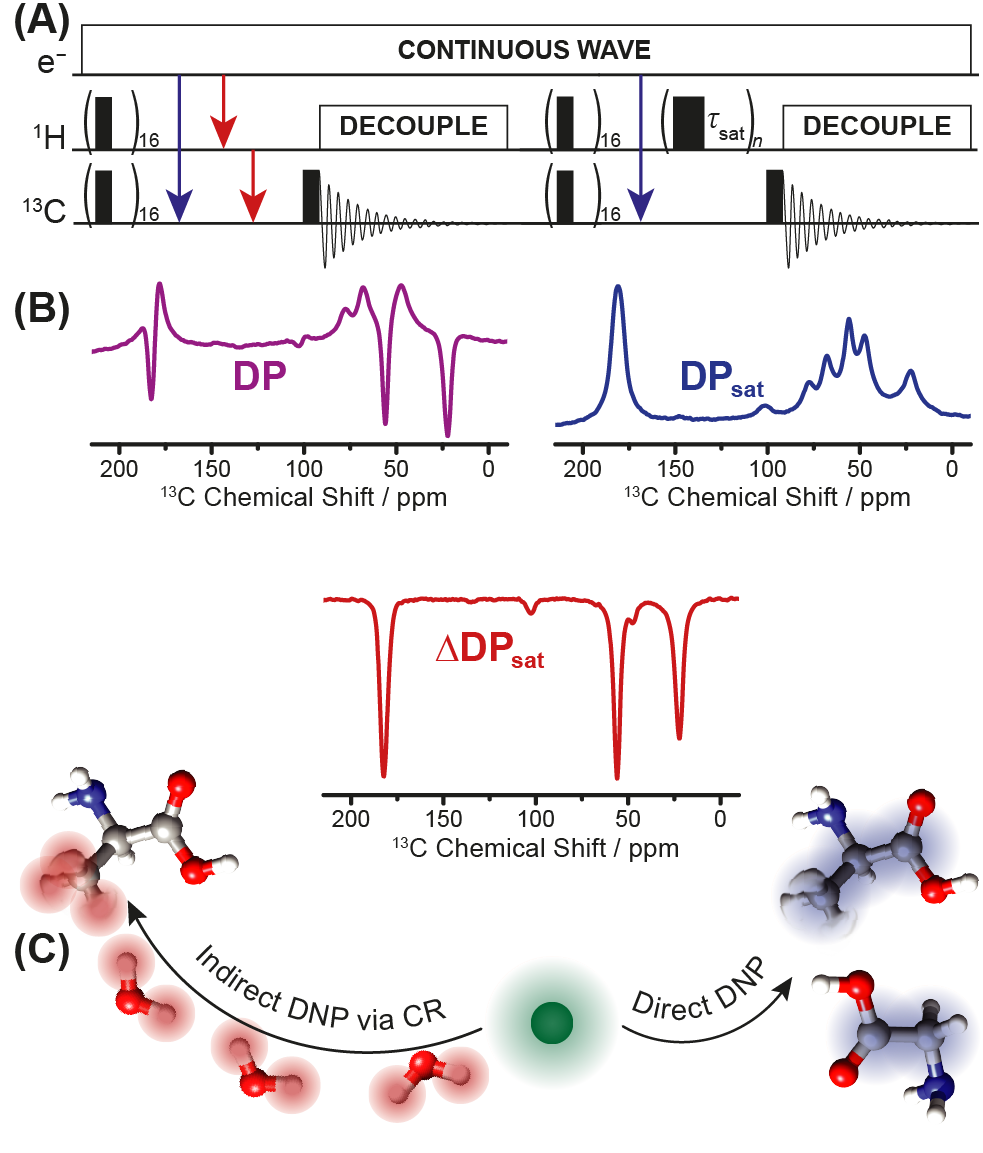
Die Übertragung der Polarisation von einem Elektronenspin auf einen Kernspin durch DNP kann auf direkte oder indirekte Weise geschehen. Bei der direkten Polarisation (Abbildung (A), blaue Pfeile) wird die Polarisation von dem Elektronspin direkt auf den Kohlenstoffspin übertragen. Bei der indirekten Polarisation geschieht die Polarisationsübertragung durch einen Zwischenschritt, bei dem die Polarisation zunächst auf die Protonen übertragen wird und erst anschließend auf die Kohlenstoff-Kerne (Abbildung (A), rote Pfeile). Sind nun in der untersuchten Probe aktive Methylgruppen vorhanden, treten sowohl der direkte Polarisationsweg, als auch der indirekte Polarisationsweg, verursacht durch die schnelle Rotation der Methylgruppen (Kern-Overhauser-Effekt), auf. Dabei resultiert der direkte Transferweg in positiven Signalen und der indirekte Transfer in negativen. Das Spektrum enthält also sowohl positive als auch negative Anteile (Abbildung (B),DP, lila). Um nun ausschließlich die Umgebung in der Nähe der Methylgruppen untersuchen zu können, müssen die positiven und die negativen Anteile des Spektrums getrennt werden können. Dazu kann eine Pulssequenz verwendet werden, bei der durch Sättigungspulse (Abbildung (A) τsat ) der indirekte Transferweg unterdrückt wird. Somit erhält man für dieses Experiment nur den positiven Anteil, welcher durch die direkte Polarisation verursacht wurde (Abbildung (B), DPsat, blau). Durch anschließende Subtraktion von Spektrum DP und DPsat erhält man ausschließlich die Anteile des indirekten Transferwegs, verursacht durch die schnelle Rotation der Methylgruppen (Abbildung (B), ΔDPsat. Dieses Spektrum enthält dann Informationen über die Umgebung der Methylgruppen. Abbildung (C) zeigt die schematische Darstellung des direkten und indirekten Polarisationsweges am Beispiel von Glycin, dass keine Methylgruppe trägt und Alanin, dass genau eine Methylgruppe trägt und damit SCREAM-aktiv ist.