DNP an RNA
DNP an Biomolekülen
Die dynamische Kernspinpolarisation (DNP) an biologischen Systemen ist experimentell herausfordernd. Zum einen wird ein typisches DNP-Experiment bei Temperaturen um 100 K durchgeführt, weswegen biomolekulare Komplexe gegen Kältedenaturierung geschützt werden müssen. Dies geschieht meist durch Zugabe von Glycerol. Dabei muss sichergestellt werden, dass Glycerol keinen Einfluss auf die Faltung und die Funktion des Biomoleküls hat. Weiterhin kommt es bei der Untersuchung von biomolekularen Komplexen oft zu spektralen Signalüberlappung, da Biomoleküle aus ähnlichen Bausteinen wie Aminosäuren (im Fall von Proteinen) oder Nukleotiden (im Fall von Nukleinsäuren) bestehen welche untereinander eine ähnliche chemische Umgebung aufweisen. Aus diesen Gründen entwickeln wir DNP-Methoden, die es erlauben, spezifisch und systematisch Teile von biomolekularen Komplexen zu untersuchen.
Ribonukleinsäuren (RNA)
Ribonukleinsäuren (RNAs) dienen nicht nur der Translation des genetischen Codes in Proteine, sondern übernehmen auch eine Vielzahl anderer Aufgaben in Zellen. Trotz des einfachen Aufbaus, bestehend aus nur vier unterschiedlichen Nukleotiden basierend auf Adenosin, Guanosin, Cytosin und Uridin, ist die Vielfalt der RNAs immens. Allgemein wird zwischen den codierenden und den nicht-codierenden RNA (ncRNA) unterschieden. Während die codierende messenger RNA (mRNA) in ein Protein umgeschrieben wird, erfüllen ncRNAs unterschiedlichste regulatorische Aufgaben in der Zelle. Einige Beispiele für ncRNAs sind die riobosomale RNA (rRNA) und die transfer RNA (tRNA), die für die Genexpression notwendig sind, die miRNA (mikroRNA), die eine wichtige Rolle bei der Chromatinumlagerungen spielt, die siRNA (small interfering RNA), welche Modifikationen an Histonen ausüben kann, die snRNA (small nuclear RNA), welche am Prozess des Spleißens in eykariotischen Organismen beteiligt ist oder die snoRNA (small nucleolar RNA), die für die Prozessierung und Modifikation von Ribonukleinsäuren notwendig ist. Oft findet man ncRNAs in Komplexen mit Proteinen, den sogenannten Ribonukleoproteinen (RNPs). Eine weitere Klasse der RNAs sind die sogenannten RNA-Aptamere. Diese werden im Labor für die Bindung eines sehr spezifischen Liganden selektiert und sind vor allem für Anwendungen in der Analytik und Krebsforschung interessant.
Das Tetracyclin-bindende Aptamer (TC-Aptamer)
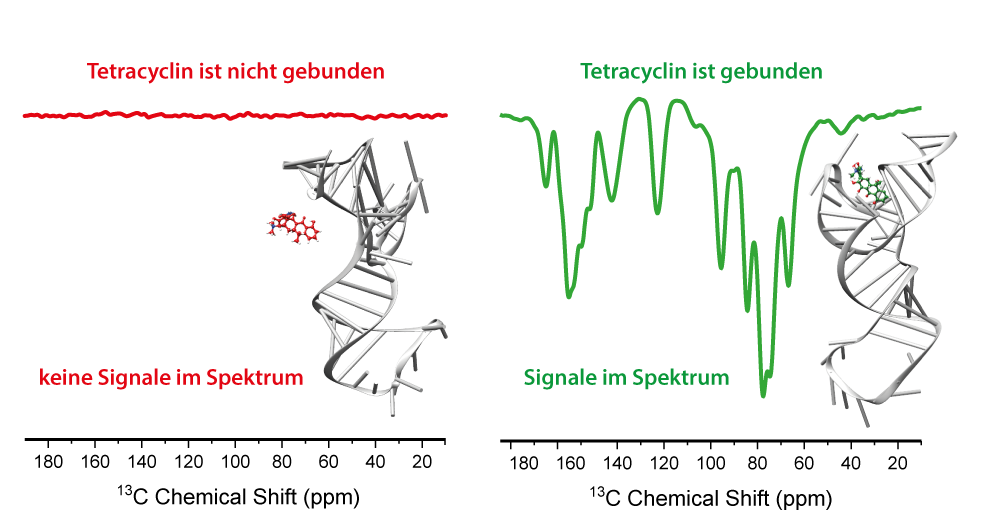
Das TC-Aptamer wurde ursprünglich synthetisiert um die Wechselwirkungen zwischen dem Antibiotikum Tetracyclin und RNA zu analysieren, konnte jedoch nach weiteren Modifikationen für die Regulation der Genexpression oder des Spleißens verwendet werden (Suess et al., Nucleic Acids Res. 2003, 31,1853). Tetracyclin besitzt drei Methylgruppen und wird mit sehr hoher Affinität von der RNA gebunden. Da die RNA in der unmodifizierten Form keine Methylgruppen trägt, ist das TC-Aptamer ein optimales System, um SCREAM-DNP zu untersuchen. Hierfür wurde Tetracyclin mit magnetisch aktiven Kohlenstoff-13-Kernen (13C) angereichert und diente somit als das methyltragende Spionmolekül (siehe Abschnitt SCREAM-DNP). Bindet dieses an die RNA, kommt es zur Ausbildung von charakteristischen NMR-Signalen des RNA-Aptamer-Komplexes, welche isoliert und damit von dem ungewünschten Hintergrund unterschieden werden kann. Werden die experimentellen Bedingungen allerdings so gewählt, dass das Aptamer das Tetracyclin-Molekül nicht binden kann, bleiben diese spezifischen Signale aus (siehe Abbildung).
Diese Studie wurde 2019 in der Angewandten Chemie (Aladin et al., Angew. Chem. Int. Ed. 2019, 58, 4863) veröffentlicht, wofür Victoria Aladin mit dem GDCh-Ernst-Preis ausgezeichnet wurde. Die Studie dient als Demonstration der Verwendung von SCREAM-DNP zur selektiven Untersuchung biomolekularer Komplexe. Durch die hohe Spezifität dieser Methode können diese Komplexe auch neben anderen Bestandteilen einer Mischung eindeutig spektral isoliert werden.
Das Hammerkopf-Ribozym (HHRz)
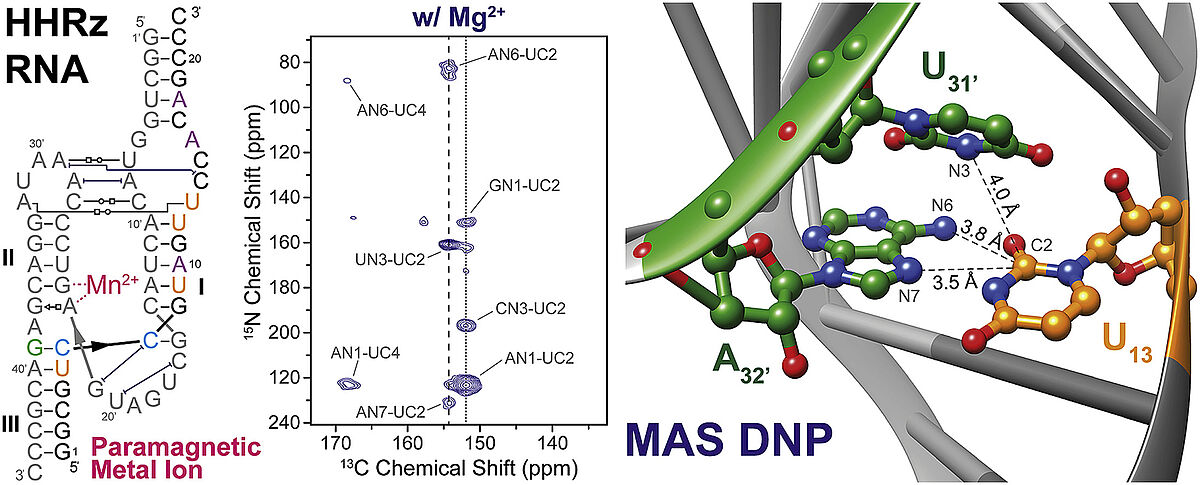
Das Hammerkopf-Ribozym ist ein struktureller Baustein eines Teils des Genoms vieler pflanzlicher Viren. Infolge der Infektion einer Wirtszelle führt das HHRz eine enzymatische Spaltungsreaktion durch, die die Replikation der viralen RNA ermöglicht. Für diese Spaltungreaktion muss eine spezifische Bindungsstelle mit divalenten Ionen (z.B. Mg2+ oder Mn2+ besetzt werden. Lange Zeit war unklar, welche strukturellen Veränderungen durch diese Koordination ausgelöst werden; allerdings konnten Martick und Scott vor einiger Zeit zeigen dass das divalente Ion wohl keinen Einfluss auf die Kristallstruktur des HHRz hat (Scott et al., Prog. Mol. Biol. Transl. Sci., 2013, 120, 1). Vor kurzem konnten wir diese Hypothese durch DNP-verstärkte MAS-NMR bestätigen. Sowohl in Anwesenheit als auch in Abwesenheit von Mg2+ zeigen sich in 2D-Korrelationsspektren (15N-13C-TEDOR) die gleichen Tertiärkontakte zwischen zwei RNA-Stammschleifen durch nicht-kanonische reverse Hoogsteen-Basenpaarung. Dies deutet darauf hin, dass auch in Lösung die spezifische Bindung von Mg2+ die strukturellen Populationen nicht signifikant beeinflusst (Daube et al., Solid State Nucl. Magn. Reson. 2019, 101, 21).
Weiterhin haben wir die Möglichkeit demonstriert, Mn2+ als Polarisationsmittel zu verwenden um ortsspezifische DNP durchzuführen. Dies ist insofern hochinteressant, dass kein exogenes Polarisationsmittel (z.B. Nitroxid) der Probenlösung hinzugefügt werden muss; DNP erfolgt mittels eines endogen gebundenen Metallions im inneren des Biomoleküls (Wenk et al., J. Biomol. NMR 2015, 63, 97).