Ortsspezifische DNP
Abstandsabhängigkeit des Elektronen-Kern-Polarisationstransfers
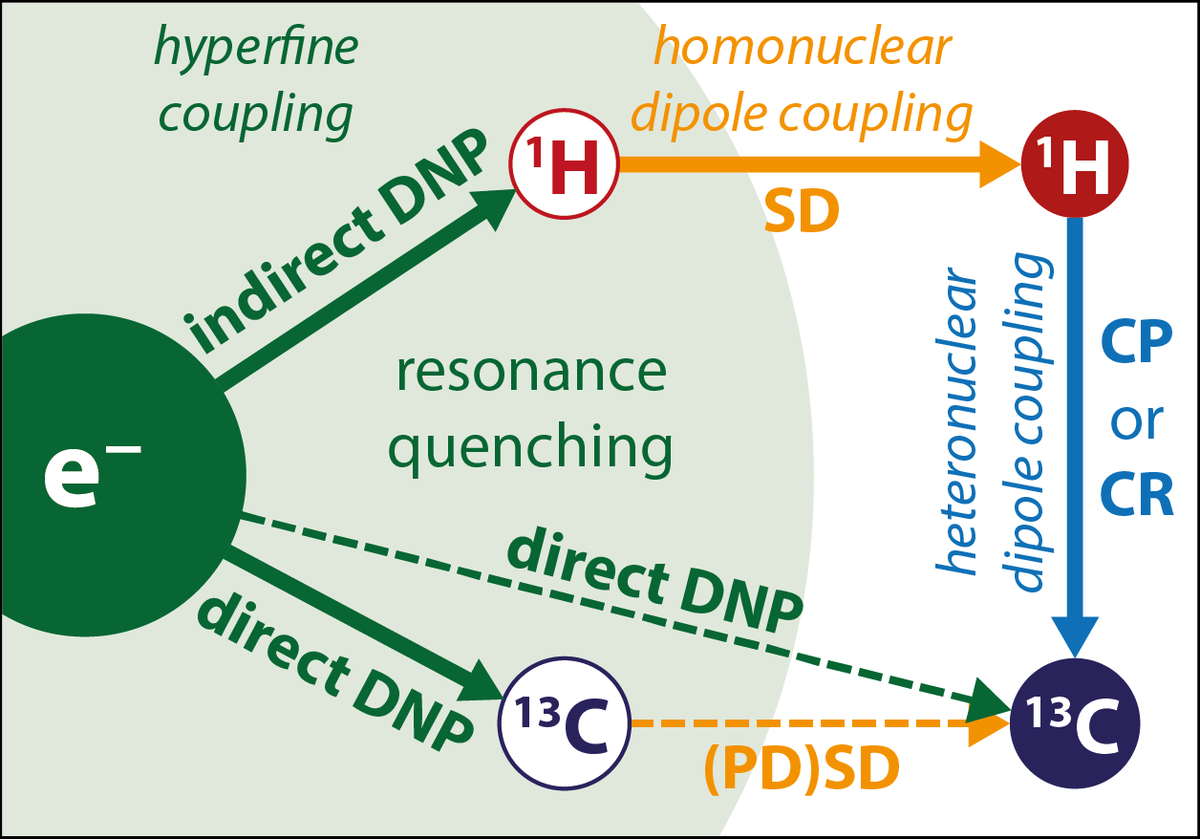
Obwohl die quantenmechanischen Grundlagen für einfache Spin-Systeme hinreichend bekannt sind, ist die Vorhersage der Distanz, über die Spin-Polarisation durch DNP übertragen werden kann, sehr schwierig. Dies ist auf der Vielzahl von unterschiedlichen Effekten begründet, die DNP beeinflussen. So ermöglicht die Hyperfein-Kopplung zwischen Elektronen- und Kernspins den Polarisations-Transfer, gleichzeitige Kopplungen zu weiteren Spins können mit diesem Transfer aber wiederum interferieren. Dadurch kommt es in einem komplexen System aus einer Vielzahl von Elektronen und Kernspins schnell zu einem unüberschaubaren Netzwerk an kompetitiven Wechselwirkungen.
Die Kenntnis über DNP-Transferraten als Funktion des Elektron-Kern-Abstands und eventueller weiterer Parameter wie Relaxationsraten und Spin-Diffusionsraten wäre ein immenser Vorteil in Blick auf zielgerichtete Anwendungen. So könnte z.B. durch gezielte Elektronenspin-Markierung von Molekülen oder Komplexen eine empfindliche Abstandsmessung durch DNP möglich sein. Ebenso wäre die selektive NMR-Detektion von markierten Komponenten einer Mischung denkbar.
Ubiquitin mit Gd-Spinlabeln
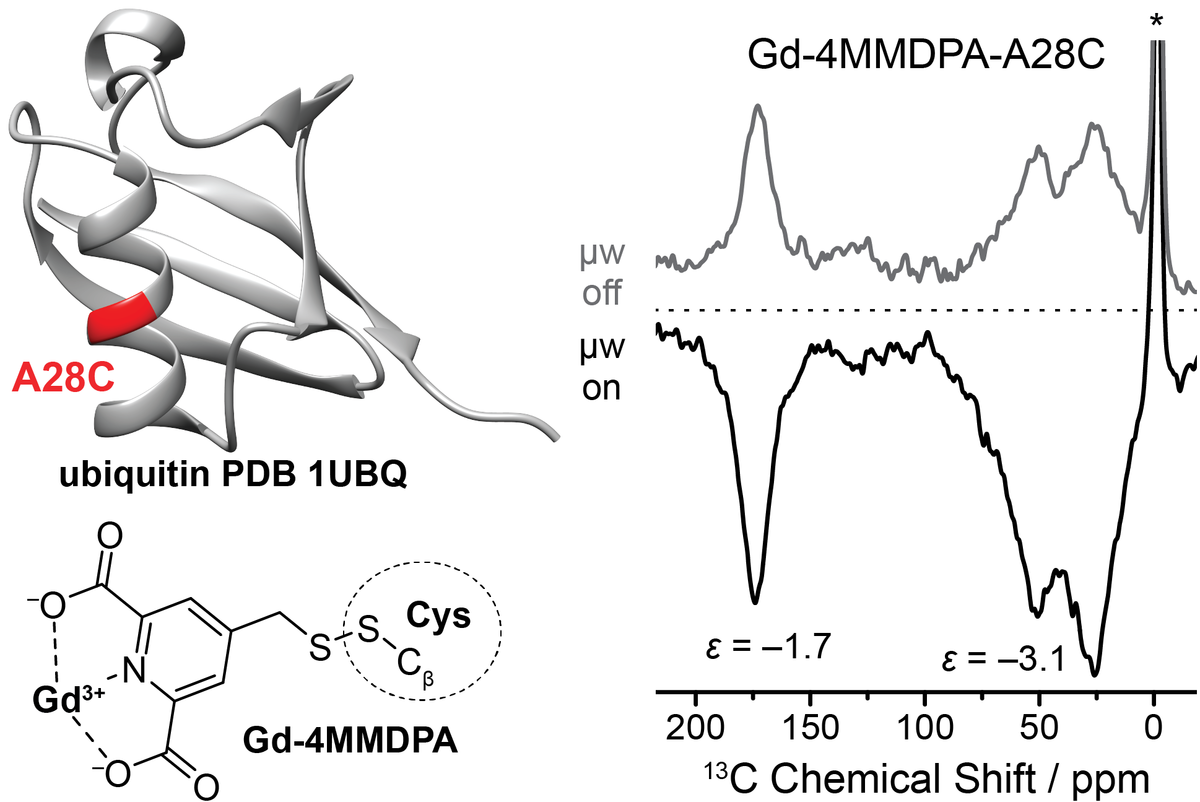
Ein Modellsystem, das wir zur Untersuchung dieser Fragestellung entwickelt haben, basiert auf Ubiquitin. Dieses allgegenwärtige Protein spielt eine wichtige Rolle beim Abbau von nicht mehr benötigten Proteinen. Bei der Proteolyse binden ein oder mehrere Ubiquitin-Moleküle an das entsprechende Protein durch kovalente Kopplung des C-Terminus an ein Lysin. Dieser Ubiquitin-Komplex wird durch das Proteasom erkannt und degradiert. Durch diese Funktion ist Ubiquitin nicht nur ein bereits sehr gut untersuchtes Modellsystem für unsere Forschung, sondern bietet auch vielfältige Möglichkeiten zur weiteren Untersuchung der Proteolyse mittels DNP.
In einer ersten Studie haben wir die Aminosäuren-Sequenz von Ubiquitin an drei unterschiedlichen Stellen verändert und an den entsprechenden Mutationsstellen zielgerichtet Spin-Marker chemisch gebunden. Diese Spin-Marker basieren auf dem Metallion Gd3+, welches mit einem Chelatkomplex fest an das Protein gebunden ist. Weiterhin konnten wir zeigen, dass diese Metallionen als DNP-Polarisationsmittel für die spezifische Hyperpolarisation von Kernspins innerhalb des Proteins verwendet werden können.
Ortsspezifische DNP in perdeuteriertem Protein
In einer aktuellen Studie untersuchen wir den Einfluss der Protonen-Konzentration auf die direkte DNP von Kernspins in Gd-DOTA-Ubiquitin. Durch Deuterierung des Proteins während der rekombinanten Expression ist die gezielte Variation des Isotopen-Verhältnisses von Protonen und Deuteronen möglich. Hierdurch lassen sich die Spin-Diffusions- sowie die Kreuzrelaxationsrate von 13C und 15N beeinflussen. Dies ermöglicht zum einen eine signifikante Vergrößerung des direkten DNP-Verstärkungsfaktors, zum anderen konnten wir zum ersten Mal zeigen, dass direkte DNP prinzipiell zur Messung von Abständen zwischen Elektronen- und Kernspins verwendet werden kann. Diese Ergebnisse werden in naher Zukunft veröffentlicht.